Revisor: Dra. Ebymar Arismendi. Servicio de Neumología Hospital Clínic de Barcelona
Artículo seleccionado:: Pauline J. M. Kuks, Tatiana Karp, Jorine E. Hartman, Monica Kraft, Salman Siddiqui,Leonardo M. Fabbri, Bianca Beghé, Klaus F. Rabe, Alberto Papi, Christopher E. Brightling, Dave Singh,Xingnan Li, Janwillem W. H. Kocks, Ulrica Scaffidi-Argentina, Huib A. M. Kerstjens, Irene H. Heijink, Simon D. Pouwels, Dirk-Jan Slebos, Maarten van den Berge. Cluster Analysis to Identify Distinct Asthma Phenotypes in the ATLANTIS Cohort. Allergy 2025; 0:1–12
Palabras clave: inflamación de la vía aérea, fenotipos de asma, análisis de clústeres, perfil de expresión génica, disfunción de la vía aérea pequeña
Resumen breve
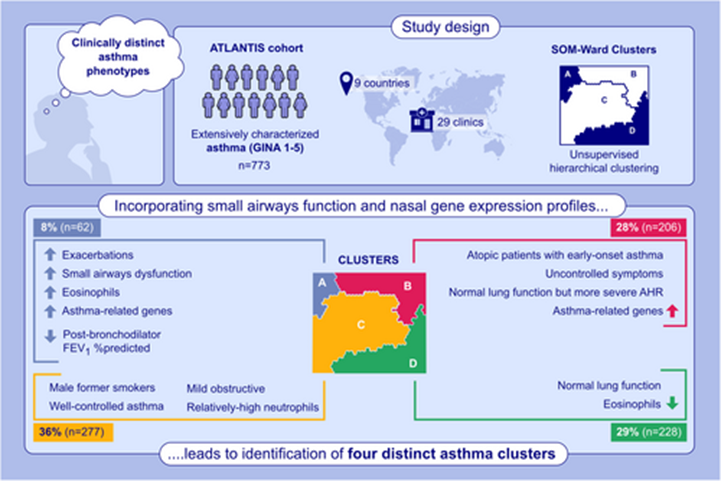
El estudio ATLANTIS incluyó a 773 adultos con asma (GINA 1–5), evaluados de forma integral con datos clínicos, función pulmonar, recuentos celulares en sangre y esputo y perfil de expresión génica nasal. Mediante un “cluster analysis” no supervisado se identificaron cuatro grupos de pacientes y se observó que la disfunción de la vía aérea pequeña (SAD) se relacionaba con mayor inflamación tipo 2, peor función pulmonar y mayor número de exacerbaciones, por lo que se propone como un rasgo clave a tener en cuenta en la estratificación del asma.
Hipótesis
La hipótesis del estudio es que la incorporación de parámetros de SAD y del perfil de expresión génica nasal en un análisis de clusters identifica fenotipos de asma clínicamente diferenciados más allá de la gravedad según GINA, y que la presencia de SAD se asocia a mayor inflamación tipo 2, peor función pulmonar y más exacerbaciones.
Métodos
En el estudio ATLANTIS se incluyeron 773 adultos con asma (18–65 años, GINA 1–5) en situación estable, reclutados entre junio de 2014 y marzo de 2017 en 29 centros de nueve países. Se realizó una caracterización detallada que incluyó datos clínicos (historia de la enfermedad, control del asma, cuestionarios validados), función pulmonar con espirometría, oscilometría de impulso y lavado de nitrógeno en múltiples respiraciones, así como tomografía computarizada torácica (TC) para la evaluación de volúmenes pulmonares y atrapamiento aéreo. Asimismo, se determinaron recuentos celulares diferenciales en sangre periférica y esputo inducido y se llevó a cabo el perfil de expresión génica en epitelio nasal mediante técnicas de transcriptómica, posteriormente resumido en módulos génicos. A partir de este amplio conjunto de variables se seleccionó un panel de 36 parámetros representativos de los dominios clínico, funcional, radiológico, inflamatorio y molecular. Con dichas variables se realizó un análisis de clusters no supervisado (Self-Organizing Map–Ward) para agrupar a los pacientes según la similitud de sus perfiles, y se compararon posteriormente los grupos obtenidos en términos de función pulmonar, inflamación y exacerbaciones con el fin de definir fenotipos de asma diferenciados.
Objetivos
Identificar fenotipos de asma clínicamente diferenciados, más allá de la gravedad según GINA, mediante un análisis de clusters no supervisado en la cohorte ATLANTIS, incluyendo de forma específica parámetros de SAD y el perfil de expresión génica nasal.
Resultados
A partir de más de 800 variables iniciales se seleccionaron 36 (clínicas, funcionales, radiológicas, inflamatorias y transcriptómicas) y se aplicó un algoritmo de mapas auto-organizados (Self-Organizing Map–Ward) de forma totalmente no supervisada.
Se identificaron cuatro clusters.
- El Cluster A (8%), “T2-alto con SAD”, agrupa a los pacientes con asma más grave (95% GINA 4–5), peor control, más exacerbaciones en 12 meses, FEV₁ y FEF25–75 más bajos, marcada afectación de la vía aérea pequeña (RV/TLC, R5–20, Scond alterados) y más remodelado y atrapamiento aéreo en la TC. Presentan eosinofilia en sangre y esputo y alta actividad de los módulos génicos 4–6 en epitelio nasal. Los autores subrayan que la SAD es un rasgo integral del asma T2-alto grave y un posible “rasgo tratable” (p. ej. formulaciones extrafinas, antileucotrienos, biológicos).
- El Cluster B (27%) corresponde a asma de inicio precoz, alérgica, con síntomas mal controlados e hiperrespuesta bronquial (HRB) más grave, pero con función pulmonar globalmente preservada y sin SAD. Los marcadores clásicos T2 (eosinófilos sanguíneos/esputo, FeNO) no están especialmente elevados, pero el epitelio nasal muestra una fuerte sobreexpresión de genes relacionados con asma (módulos 4–6, incluidos CLCA1 y POSTN). Esto sugiere actividad inflamatoria relevante no captada por marcadores rutinarios y un posible infratratamiento (menor uso de corticoides inhalados). Los autores plantean que este subgrupo podría tener riesgo futuro de progresión y beneficiarse de una intensificación terapéutica precoz.
- El Cluster C (36%) está formado en su mayoría por varones exfumadores, con mayor carga tabáquica, asma bien controlada pero obstrucción leve (FEV₁ y FEF25–75 reducidos frente a B y D) y niveles relativamente altos de neutrófilos en esputo. Representa un fenotipo “asma leve obstructiva, exfumadores con neutrofilia relativa”. Aunque trabajos previos en ATLANTIS no habían asociado neutrofilia en esputo a un fenotipo clínico claro, aquí sí se identifica un subgrupo con peor función pulmonar y más exposición tabáquica en el que los neutrófilos podrían desempeñar un papel.
- El Cluster D (29%) es un fenotipo de “asma leve con baja inflamación”: síntomas bien controlados, función prácticamente normal tanto en vía aérea grande como pequeña, recuentos bajos de eosinófilos, neutrófilos y otras células en sangre y esputo. A nivel nasal, los módulos génicos 1–3 (genes habitualmente infrarregulados en asma) aparecen “normalizados” y los módulos 4–6 se encuentran infraexpresados, configurando un transcriptoma similar al de sujetos sanos.
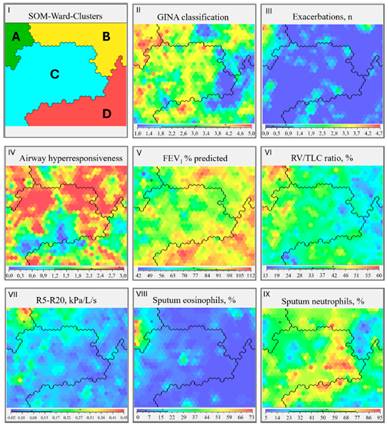
La figura muestra cómo el algoritmo de cluster analysis distribuye a los 773 pacientes en un mapa donde los sujetos con perfiles similares se agrupan formando cuatro clústeres (A–D). Sobre ese mapa se superponen distintos parámetros clínicos y fisiopatológicos (gravedad GINA, exacerbaciones, AHR, FEV₁, RV/TLC, R5–R20 y porcentajes de eosinófilos y neutrófilos en esputo), codificados por colores, para visualizar qué rasgos caracterizan cada clúster.
En conjunto, el trabajo muestra que incorporar medidas de SAD y transcriptómica nasal permite mejorar la estratificación fenotípica más allá de lo que ofrecen GINA, la espirometría y los biomarcadores T2 convencionales. La presencia de SAD se asocia de forma consistente con inflamación tipo 2 marcada, peor función pulmonar y mayor número de exacerbaciones, apoyando su consideración como rasgo clínico clave en asma. Por otra parte, el cluster de inicio precoz alérgico con función pulmonar preservada pero HRB marcada y alta expresión génica T2 podría representar un grupo “aparentemente leve” pero con riesgo futuro, donde la valoración de HRB y marcadores moleculares aportaría información pronóstica adicional. Entre las limitaciones, los autores señalan el seguimiento relativamente corto (1 año), la ausencia de reevaluación de todas las variables clave para estudiar la estabilidad de los clusters, la falta de validación externa y la ausencia de información sistemática sobre comorbilidades.
Mensajes claves
Este estudio describe cuatro clústeres de asma diferenciados:
A) Asma con Inflamación T2-alto y SAD;
B) Asma de inicio precoz con HRB marcada;
C) Asma leve con obstrucción en exfumadores con neutrofilia relativa;
D) Asma leve con baja inflamación.
Los autores destacan que evaluar la SAD y la HRB es clave, incluso en pacientes con función pulmonar relativamente conservada, y que la información transcriptómica en el asma de inicio precoz ayuda a entender mejor los mecanismos de la enfermedad. En conjunto, subrayan la necesidad de integrar datos clínicos y moleculares para una fenotipificación más precisa y tratamientos más personalizados.







